Комплект багатоядерної РНК-Seq TIANSeq (освітлений)
Особливості
■ Хороша однорідність послідовності: висока точність ампліфікації ПЛР та відсутність зміщення бази.
■ Висока ефективність конвертації бібліотек: Високоефективну конструкцію бібліотеки можна забезпечити для зразків мРНК 1 нг.
■ Швидка робота: На весь процес побудови бібліотеки потрібно всього 5,5 годин.
Специфікація
Тип: Підготовка бібліотеки спрямованої РНК РНК
Зразок: Загальна РНК
Ціль: мРНК, lncRNA
Початковий зразок введення: Загальна кількість зразків РНК становить 10 нг-1 мкг, а зразка мРНК-всього 1 нг
Час роботи: 5,5-6,5 години
Нижчі потоки програм: Послідовність на платформі illumina.
Усі продукти можна налаштувати для ODM/OEM. Для деталей,будь ласка, натисніть Індивідуальне обслуговування (ODM/OEM)
Порівняння врожаю бібліотек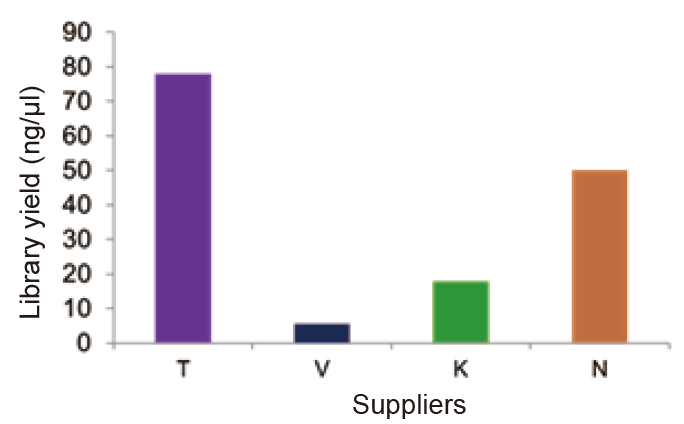 |
Малюнок 1. Бібліотека РНК з 1 мкг загальної РНК людської клітини 293T була побудована з використанням набору TIANSeq Stranded RNASeq та відповідних продуктів від постачальників V, K та N. Результат показує, що комплект із багатоядерної РНК-Seq TIANSeq має значно вищий вихід, ніж у постачальника V , K та N. |
Рівномірне покриття транскриптомів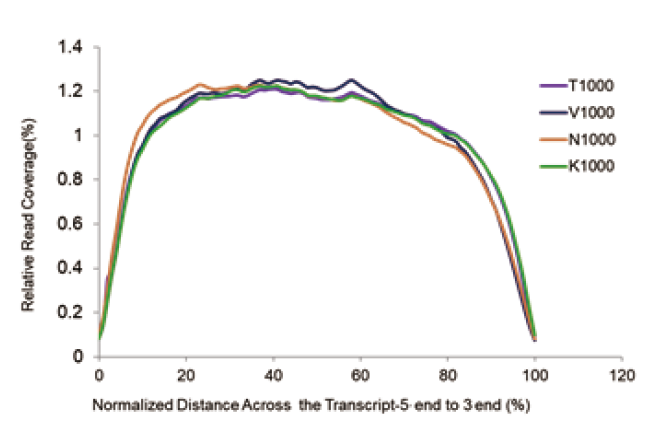 |
Малюнок 2. Бібліотека РНК з 1 мкг загальної РНК людської клітини 293Т була побудована з використанням набору TIANSeq Stranded RNASeq та відповідних продуктів від постачальників V, K та N. Результат показує, що набір TIANSeq Stranded RNASeq мав дуже рівномірне покриття від 5 'до 3' кінець, який був кращим за Постачальника V, а продуктивність була подібною до продукції Постачальника N та K. |
Широкий діапазон застосування вибіркового введення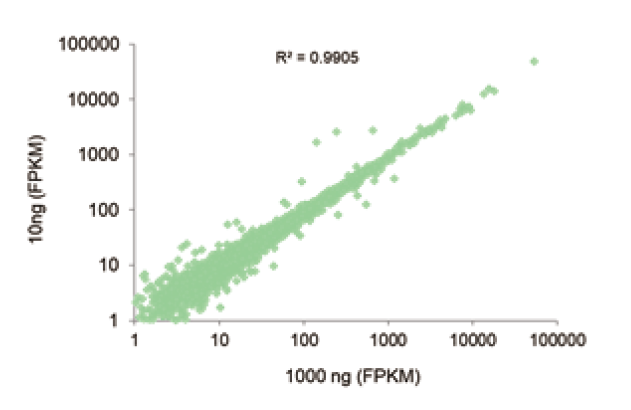 |
Малюнок 3. Комплект багатоядерної РНК-Seq TIANSeq сумісний із загальною вхідною РНК 10 нг-1 мкг, а бібліотека, побудована під різними вхідними зразками, має високу послідовність. Результат показує, що коефіцієнт кореляції експресії генів між бібліотеками досягав вище 0,99 при загальному введенні РНК 10 нг та 1 мкг клітини 293Т, а узгодженість даних була високою. |
В даний час високопродуктивна технологія секвенування в основному базується на технології секвенування нового покоління. Оскільки довжина читання технології послідовності наступного покоління обмежена, ми повинні розділити послідовність повної довжини на невеликі бібліотеки фрагментів для послідовності. Відповідно до потреб різних експериментів з секвенуванням, ми зазвичай обираємо одностороннє або двостороннє. В даний час фрагменти ДНК бібліотеки секвенування наступного покоління зазвичай розподіляються в діапазоні 200-800 п.н.

а) ДНК поганої якості та містить інгібітори. Використовуйте високоякісні зразки ДНК, щоб уникнути пригнічення активності ферментів.
б) Кількість зразка ДНК недостатня при використанні методу без ПЛР для побудови бібліотеки ДНК. Коли вхід фрагментованої ДНК перевищує 50 нг, робочий процес без ПЛР може бути вибірково здійснений під час процесу побудови бібліотеки. Якщо номер копії бібліотеки занадто низький, щоб її можна було безпосередньо секвенувати, бібліотеку ДНК можна ампліфікувати за допомогою ПЛР після лігування адаптера.
в) Забруднення РНК призводить до неточної початкової кількісної оцінки ДНК Забруднення РНК може існувати в процесі очищення геномної ДНК, що може призвести до неточної кількісної оцінки ДНК та недостатнього завантаження ДНК під час побудови бібліотеки. РНК можна видалити шляхом обробки РНКазою.
А-1
а) З'являються невеликі фрагменти (60 п.н.-120 п.н.). Очищення за допомогою магнітних кульок Agencourt AMPure XP може ефективно видалити ці фрагменти адаптера та забезпечити якість послідовності.
б) Великі фрагменти з'являються в бібліотеці після ампліфікації ПЛР. Розмір бібліотечного фрагмента ДНК збільшиться на 120 п.н. після лігування адаптера. Якщо фрагмент ДНК збільшується більш ніж на 120 п.н. після перев'язки адаптера, це може бути викликано аномальною ампліфікацією фрагмента надмірною ампліфікацією ПЛР. Зменшення кількості циклів ПЛР може запобігти ситуації.
c) Аномальний розмір бібліотечних фрагментів ДНК після лігування адаптера Довжина адаптера в цьому наборі становить 60 п.н. Коли два кінці фрагмента будуть перев’язані до адаптерів, довжина збільшиться лише на 120 п.н. Якщо ви використовуєте адаптер, відмінний від того, що надається цим набором, зверніться до постачальника, щоб надати відповідну інформацію, таку як довжина адаптера. Будь ласка, переконайтеся, що робочий процес та робота експерименту відповідають крокам, описаним у посібнику.
d) Аномальний розмір фрагмента ДНК до перев'язки адаптера Причиною цієї проблеми можуть бути неправильні умови реакції під час фрагментації ДНК. Для різного введення ДНК слід використовувати різний час реакції. Якщо вхід ДНК перевищує 10 нг, ми рекомендуємо вибрати час реакції 12 хвилин як час початку оптимізації, а розмір фрагмента, отриманого в цей час, знаходиться переважно в діапазоні 300-500 п.н. Користувачі можуть збільшувати або зменшувати довжину фрагментів ДНК протягом 2-4 хв відповідно до власних вимог для оптимізації фрагментів ДНК з необхідним розміром.
А-2
а) Час фрагментації не оптимізовано Якщо фрагментована ДНК занадто мала або занадто велика, будь ласка, зверніться до Настанов щодо вибору часу фрагментації, наданих в інструкції, щоб визначити час реакції, і використовуйте цей момент часу як контроль, додатково встановіть реакційну систему, щоб подовжити або скоротити 3 хвилини для більш точного регулювання часу фрагментації.
А-3
Аномальний розподіл ДНК за розміром після обробки фрагментацією
а) Неправильний метод розморожування реагенту дроблення, або реагент не повністю перемішується після розморожування. Розморозьте 5 -кратний реагент для фрагментації ферментної суміші на льоду. Після розморожування рівномірно перемішайте реагент, обережно натискаючи дно пробірки. Не вихровуйте реагент!
б) Вхідний зразок ДНК містить ЕДТА або інші забруднювачі. Виснаження іонів солі та хелатуючих агентів на етапі очищення ДНК особливо важливо для успіху експерименту. Якщо ДНК розчиняється в 1 × TE, використовуйте метод, наведений в інструкції, для фрагментації. Якщо концентрація ЕДТА в розчині невизначена, рекомендується очистити ДНК і розчинити її в деіонізованій воді для подальшої реакції.
в) Неточне початкове кількісне визначення ДНК Розмір фрагментованої ДНК тісно пов'язаний з кількістю введеної ДНК. Перед обробкою фрагментацією для визначення точної кількості ДНК в реакційній системі необхідна точна кількісна оцінка ДНК за допомогою Qubit, Picogreen та інших методів.
d) Підготовка реакційної системи не відповідає інструкціям. Підготовку фрагментованої реакційної системи слід проводити на льоду строго відповідно до інструкцій. Для забезпечення найкращого ефекту всі компоненти реакції слід покласти на лід і підготувати реакційну систему після повного охолодження. Після завершення підготовки, будь ласка, проведіть пальцем або піпеткою, щоб ретельно перемішати. Не вихровіть!
1. Неправильний метод змішування (вихровий, сильні коливання тощо) спричинить аномальний розподіл фрагментів бібліотеки (як показано на наступному малюнку), що вплине на якість бібліотеки. Тому, готуючи реакційний розчин Fragmentation Mix, будь ласка, акуратно піпетуйте вгору і вниз, щоб перемішати, або використовуйте кінчик пальця, щоб гортати і рівномірно перемішувати. Будьте обережні, щоб не змішати з вихором.

2. Для побудови бібліотеки необхідно використовувати ДНК високої чистоти
■ Хороша цілісність ДНК: діапазон електрофорезу становить більше 30 кб, без хвостів
■ OD260/230:> 1.5
■ OD260/280: 1,7-1,9
3. Кількість введеної ДНК має бути точною. Для кількісної оцінки ДНК пропонується використовувати методи Qubit та PicoGreen, а не Nanodrop.
4. Необхідно визначити вміст ЕДТА у розчині ДНК. ЕДТА має великий вплив на реакцію фрагментації. Якщо вміст ЕДТА високий, перед наступним тестом необхідно провести очищення ДНК.
5. Розчин реакції дроблення необхідно готувати на льоду. Процес дроблення чутливий до температури та часу реакції (особливо після додавання підсилювача). Щоб забезпечити точність часу реакції, підготуйте реакційну систему на льоду.
6. Час реакції фрагментації повинен бути точним. Час реакції на етапі фрагментації безпосередньо впливатиме на розмір продуктів -фрагментів, тим самим впливаючи на розподіл розмірів фрагментів ДНК у бібліотеці.
1. Який тип зразка застосовується до цього набору?
Відповідним типом зразка цього набору може бути загальна РНК або очищена мРНК з хорошою цілісністю РНК. Якщо для побудови бібліотеки використовується загальна РНК, для видалення рРНК спочатку рекомендується використовувати комплект для виснаження рРНК (Cat#4992363/4992364/4992391).
2. Чи можна використати зразки FFPE для створення бібліотеки за допомогою цього набору?
МРНК у зразках FFPE буде деградувати до певної міри з відносно поганою цілісністю. При використанні цього набору для побудови бібліотеки рекомендується оптимізувати час фрагментації (скоротити час фрагментації або не виконувати фрагментацію).
3. З огляду на крок вибору розміру, наведений у посібнику з виробу, що може спричинити незначне відхилення вставленого сегмента?
Вибір розміру повинен здійснюватись у суворій відповідності з кроком вибору розміру в цьому посібнику з виробу. Якщо є відхилення, причина може полягати в тому, що магнітні намистинки не збалансовані до кімнатної температури або не змішані повністю, піпетка не точна або рідина залишилася в наконечнику. Для експерименту рекомендується використовувати наконечники з низькою адсорбцією.
4. Вибір адаптерів у будівництві бібліотек
Конструкторський комплект бібліотеки не містить реагента-адаптера, і рекомендується використовувати цей комплект разом з адаптером TIANSeq з одним індексом (Illumina) (4992641/4992642/4992378).
5. КК бібліотеки
Кількісне виявлення бібліотеки: Qubit і qPCR використовуються для визначення масової концентрації та молярної концентрації бібліотеки відповідно. Операція суворо відповідає інструкції до виробу. Концентрація бібліотеки, як правило, відповідає вимогам секвенування NGS. Виявлення діапазону розповсюдження бібліотек: Використання біоаналізатора Agilent 2100 для виявлення діапазону розповсюдження бібліотек.
6. Вибір номера циклу підсилення
Відповідно до інструкцій, кількість ПЛР-циклів становить 6-12, а кількість необхідних ПЛР-циклів слід вибирати відповідно до введеного зразка. У високопродуктивних бібліотеках перевищення зазвичай відбувається в різному ступені, що проявляється дещо більшим піком після піку цільового діапазону при виявленні біоаналізатора Agilent 2100, або виявлена концентрація Qubit нижче, ніж у qPCR. Помірне надмірне посилення - це нормальне явище, яке не впливає на послідовність бібліотек та подальший аналіз даних.
7. Спайки з'являються в профілі виявлення біоаналізатора Agilent 2100
Поява спайків у виявленні біоаналізатора Agilent 2100 пояснюється нерівномірною фрагментацією зразків, де буде більше фрагментів певного розміру, і це стане більш очевидним після збагачення ПЛР. У цьому випадку пропонується не виконувати вибір розміру, тобто встановити умову фрагментації на 94 ° C протягом 15 хвилин інкубування, де розподіл фрагментів невеликий і концентрований, а однорідність можна покращити.
Категорії товарів
ЧОМУ ВИБРАТИ НАС
З моменту свого заснування наша фабрика розробляє першокласну продукцію з дотриманням принципу
якість насамперед. Наші вироби завоювали чудову репутацію в галузі та цінні довіри серед нових і старих клієнтів.








